RESUMEN
En la presente investigación se estudian alteraciones en la expresión de algunos microRNAs (miRNAs) en la carcinogénesis cervical, las cuales parecen estar relacionados con la metilación aberrante de sus promotores. Se evaluó la frecuencia de metilación del promotor del microRNA-218-1 (miR-218-1) en lesiones escamosas intraepiteliales de bajo grado (LEIBG) y cáncer cervical (CC) con virus del papiloma humano tipo 16 (VPH 16). Se incluyeron 16 muestras de LEIBG y 16 de CC con VPH 16. La metilación del promotor de miR-218-1 se determinó con EpiTect® Methyl II PCR Array (QIAGEN). El promotor de miR-218-1 se encontró metilado en un 43.75% (7/16) en CC y en un 6.25% (1/16) en LEIBG (p = 0.0007). El promotor de miR-218-1 estuvo metilado en un mayor porcentaje en muestras de CC que en LEIBG. Es posible que la metilación del promotor de miR-218-1 sea un mecanismo epigenético implicado en su expresión aberrante en CC.
ABSTRACT
The alterations in the expression of some microRNAs (miRNAs) in cervical carcinogenesis seems to be related to the aberrant methylation of their promoters. This study evaluates the methylation frequencies of microRNA-218-1 (miR-218-1)’s promoter in low grade squamous intraepithelial lesions (LGSIL) and cervical cancer (CC) with Human Papillomavirus type 16 (HPV-16). In addition,16 samples with LGSIL and 16 with CC were included. The methylation status was performed with qPCR Epitect Methyl II Arrays. The promoter of miR-218-1 was found methylated in 43.75% (7/16) in CC and 6.25% (1/16) in LGSIL (p = 0.037). The promoter of miR-218-1 was found methylated in a higher percentage in CC samples than in LGSIL. It is possible that promoter methylation of miR-218-1 to be an epigenetic mechanism involved in their aberrant expression in CC.
INTRODUCCIÓN
El cáncer cervical (CC) es una enfermedad compleja que involucra la expresión anormal de oncogenes y genes supresores de tumor (Peng et al., 2012). La infección persistente por virus del papiloma humano de alto riesgo (VPH-AR) como el VPH 16 se considera el factor causal más importante para desarrollar CC (Zheng & Wang, 2011). La actividad transformante del VPH-AR se explica principalmente por la actividad de sus oncoproteínas E6 y E7. Éstas regulan importantes procesos biológicos celulares, como la apoptosis, proliferación celular, estabilidad cromosómica, diferenciación celular, la respuesta inmunológica y mecanismos epigenéticos como la metilación de genes y expresión de microRNAs (miRNAs), entre otros (Lizano-Soberón, 2009).
Los miRNAs son pequeñas moléculas de Ácido Ribonucleico (ARN) monocatenario de longitud de 20-23 nucleótidos (nt) que se localizan tanto en regiones intragénicas como intergénicas (Gil, 2012; Palmero et al., 2011). Los genes de miRNAs constituyen el 1%-2% de los genes conocidos en los eucariotas (John et al., 2004). Se considera que los miRNAs son reguladores de importantes procesos biológicos, tales como el crecimiento celular, la apoptosis, la eliminación de la infección viral y el desarrollo del cáncer (Hu et al., 2010).
Las alteraciones genéticas y epigenéticas son algunos de los mecanismos responsables de la desregulación de los miRNAs en cáncer (Li, Cao, Zhang, Yin & Xu, 2009). En el cáncer, los cambios epigenéticos de los miRNAs, tales como la metilación del Ácido Desoxirribonucleico (ADN) y las modificaciones que sufren las histonas, son importantes en la regulación de su expresión (Li et al., 2009). La disminución en la expresión de genes supresores de tumor en cáncer está vinculado con la hipermetilación de regiones ricas en citosinas adyacentes a guaninas (islas CpG) en sus promotores (Esteller, 2007). El microRNA-218 (miR-218) se encuentra frecuentemente desregulado en CC (Yamamoto et al., 2013). El silenciamiento de miR-218 en CC se ha relacionado con la infección por virus del papiloma humano (VPH), sin embargo la expresión disminuida de miR-218 se ha observado tanto en líneas celulares VPH positivas como en líneas celulares negativas a VPH (Yamamoto et al., 2013). En cáncer oral, la disminución de la expresión de miR-218 se ha relacionado con la hipermetilación del ADN (Yamamoto et al., 2013). Debido a estos hallazgos es posible que la metilación aberrante de promotores de miRNAs sea responsable de la expresión alterada de algunos genes de miRNAs con funciones supresoras de tumor, por lo que el objetivo de este estudio fue evaluar el estado de metilación del promotor de miR-218-1 en muestras de lesiones escamosas intraepiteliales de bajo grado (LEIBG) y CC con infección por VPH 16.
MATERIALES Y MÉTODOS
Muestras de pacientes
Se realizó la captación de biopsias de pacientes con diagnóstico de CC que acudieron al Instituto Estatal de Cancerología Dr. Arturo Beltrán Ortega, en Acapulco, Guerrero, durante el periodo de mayo a noviembre de 2013; durante el mismo periodo se captaron muestras de pacientes con diagnóstico de LEIBG en el Laboratorio de Citopatología e Inmunohistoquímica de la Unidad Académica de Ciencias Químico-Biológicas de la Universidad Autónoma de Guerrero (UACQB-UAG), Chilpancingo, Guerrero. Sólo se incluyeron en el estudio las pacientes con diagnóstico de LEIBG y CC con infección por VPH 16, y que aceptaron participar en el estudio y firmar su consentimiento informado. El ADN obtenido de las muestras se almacenó a -20 °C hasta su procesamiento.
Restricción del ADN de muestras de pacientes con LEIBG y CC
Previo al análisis de metilación, el ADN total de cada muestra fue digerido utilizando EpiTect II DNA Methylation Enzyme Kit (QIAGEN). El kit contiene dos enzimas, una enzima sensible a metilación (Enzima A) que digiere ADN desmetilado y una enzima dependiente de metilación (Enzima B) que digiere ADN metilado. Se preparó una reacción que consistió de 1 μg de ADN total, 26 μl de buffer de digestión (5X) y se llevó a un volumen final de 120 μl utilizando agua libre de DNasas y RNasas. Posteriormente, se repartieron 28 μl en cuatro tubos eppendorf para obtener cuatro reacciones de restricción; una reacción en la cual no se le agregaron enzimas de restricción (Mo) (que representó la cantidad total de ADN), una reacción con 1 μl de enzima sensible a metilación “A” (Ms), una reacción con 1 μl de enzima dependiente de metilación “B” (Md), y una reacción de doble digestión en la cual se agregaron 1 μl de enzima A y 1 μl de enzima B (Msd) y se llevaron a un volumen final de 30 μl. Las cuatro reacciones se incubaron a 37 °C durante toda la noche. Posteriormente las enzimas fueron inactivadas a 65 °C por 20 min y almacenadas a -20 °C hasta su utilización.
Reacción en cadena de la polimerasa en tiempo real (qPCR) para determinar el estado de metilación del promotor de miR-218-1
El estado de metilación del promotor de miR-218-1 se determinó utilizando Human Cancer miRNA EpiTect Methyl II Signature PCR Array (QIAGEN, Maryland USA). No se requiere conversión del ADN con bisulfito de sodio. Después de la digestión, el ADN residual se cuantificó mediante Applied Biosystems 7500 Real-Time PCR Systems (qPCR), utilizando cebadores que flanquean la región promotora del gen de miR-218-1 (EpiTect Methyl II PCR Primer Assay for Human hsa-mir-218-1 CpG Island 190453-QIAGEN). Se realizaron por muestra cuatro reacciones una para cada producto de digestión (Mo, Ms, Md y Msd) agregando 30 μl del producto de digestión, 330 μl de master mix para qPCR (SYBR® Green ROX qPCR Mastermix-QIAGEN) y 300 μl de agua libre de DNasas y RNasas (volumen final 660 μl). Se dispensaron 25 μl en una placa de qPCR que contenía los cebadores para el promotor de miR-218-1. El programa de amplificación que se utilizó fue 95 °C × 10 min para activar la DNA polimerasa, 90 °C × 30 s y 72 °C × 1 min (tres ciclos), 97 °C × 15 s y 72 °C × 1 min (40 ciclos).
Análisis de metilación del ADN
Los niveles de metilación del ADN fueron determinados de acuerdo con las instrucciones del software EpiTect Methyl DNA Methylation PCR Data Analysis (QIAGEN, 2013).
Detección y tipificación de VPH 16
La detección del VPH 16 se llevó a cabo utilizando el kit INNO LiPA genotyping Extra (Innogenetics, Barcelona, España), siguiendo las indicaciones de la casa comercial. Amplificando un segmento de 65 pb de la región L1 del genoma viral, que permite detectar por lo menos 54 tipos distintos de VPH, entre ellos el VPH 16.
RESULTADOS
Se incluyeron en el estudio 32 muestras de mujeres, divididas en dos estratos; 16 muestras de LEIBG con infección por VPH 16 y 16 muestras de CaCU con infección por VPH 16. La edad media para el grupo de LEIBG fue de 33 años y para CC de 52.71 años. Al analizar la frecuencia de metilación del promotor de miR-218-1 fue mayor en CC (VPH 16+), encontrándose en un 43.75% (7/16) de las muestras en comparación con las muestras de pacientes con LEIBG (VPH 16+) que se encontró en un 6.25% (1/16).
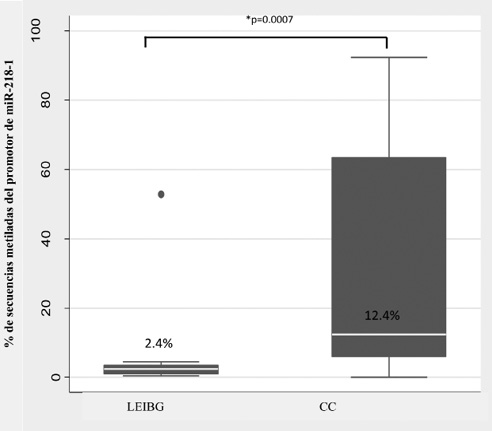
Por otra parte, la comparación del porcentaje de secuencias de ADN metiladas en ambos grupos de estudio a través de la prueba no paramétrica de Mann-Whitney arrojó diferencias estadísticamente significativas (p = 0.0007) entre CC y LEIBG, encontrándose un porcentaje mayor de secuencias metiladas en CC que en LEIBG (figura 1).
|
| |
 |
| |
Figura 1. Comparación de secuencias metiladas del promotor de miR-218-1 en muestras de pacientes con LEIGB y CC con infección por VPH 16 (*Prueba de Mann-Whitney).
Fuente: Elaboración propia. |
 |
Figura 1. Comparación de secuencias metiladas del promotor de miR-218-1 en muestras de pacientes con LEIGB y CC con infección por VPH 16 (*Prueba de Mann-Whitney).
Fuente: Elaboración propia. Close |
DISCUSIÓN
Los miRNAs desempeñan un papel importante en la carcinogénesis cervical, desde la infección por el VPH hasta la progresión a cáncer. Alteraciones en el perfil de expresión de miRNAs han sido observadas en cáncer cervical y en lesiones precursoras inducidas por VPH en comparación con tejido normal sugiriendo su participación en la carcinogénesis cervical (Zheng & Wang, 2011).
Defectos en la expresión de miRNAs han sido asociados con fallas en su regulación pos-transcripcional y/o represión transcripcional relacionados principalmente a: 1) alteraciones genéticas como deleciones, amplificaciones y mutaciones puntuales, y 2) alteraciones epigenéticas como modificación de histonas y metilación aberrante del ADN, mecanismos que pueden verse favorecidos por los VPH-AR, como el VPH 16 (Lujambio et al., 2007).
En este estudio se encontró que en pacientes con CC es más frecuente encontrar metilado el promotor de miR-218-1 en comparación con pacientes con LEIBG, además de que estos pacientes presentaron un porcentaje mayor de secuencias de ADN metiladas. En CC no existen reportes del estado de metilación del promotor de miR-218, sin embargo, la regulación epigenética por metilación de miR-218 ha sido reportada. En cáncer oral la disminución de la expresión de miR-218 por hipermetilación del ADN ha sido reportada (Yamamoto et al., 2013). Por otra parte, se ha reportado que miR-218-1 es un supresor de tumor y se encuentra disminuido en cáncer. Estudios en CC reportan que la expresión de miR-218 se encuentra desregulada (Martínez et al., 2008) y que su sobreexpresión inhibe la invasión y migración celular (Tie et al., 2010). Los resultados obtenidos en este estudio pueden sugerir que posiblemente la expresión de miR-218-1 esté silenciada por metilación de su promotor en CC y que la metilación aberrante de su promotor esté involucrada en la patogénesis del CC.
AGRADECIMIENTOS
La investigación se realizó con financiamiento del Consejo Nacional de Ciencias y Tecnología (Conacyt), convocatoria 2012 de Ciencia Básica, y con financiamiento de la Universidad Autónoma de Guerrero, Convocatoria 2013, por tal motivo se les extiende un sincero agradecimiento.
REFERENCES
Esteller, M. (2007). Epigenetic gene silencing in cancer: the DNA hypermethylome. Human Molecular Genetics, 16(1), 50-59.
Gil, G. L. (2012). MicroRNAs: nuevos marcadores de interés en oncología. Revista Eubacteria, 28(1), 1-4.
Hu, X., Schwarz, J. K., Lewis, J. S. Jr., Huettner, P. C., Rader, J. S., Deasy, J. O., Grigsby P. W. & Wang, X. (2010). A microRNA expression signature for cervical cancer prognosis. Cancer Research, 70(4), 1441-1448.
John, B., Enright, A. J., Aravin, A., Tuschl, T., Sander, C. & Marks, D. S. (2004). Human MicroRNA targets. PLoS Biology, 2(11), 1862- 1879.
Li, M., Cao, L., Zhang, H., Yin, Y. & Xu, X. (2009). Expression of 6 microRNAs in prostate cancer and its significance. Clinical Oncology Cancer Research, 6(1), 21-28.
Lizano-Soberón, M. (2009). Infección por virus del papiloma humano: epidemiología, historia natural y carcinogénesis. Revista del Instituto Nacional de Cancerología, 4(1), 205-216.
Lujambio, A., Ropero, S., Ballestar, E., Fraga, M. F., Cerrato, C., Setién, F., Casado, S., Suarez-Gauthier, A., Sanchez-Cespedes, M., Git, A., Spiteri, I., Das, P. P., Caldas, C., Miska, E. & Esteller, M. (2007). Genetic unmasking of an epigenetically silenced microRNA in human cancer cells. Cancer Research, 67(4), 1424-1429.
Martínez, I., Gardiner, A. S., Board, K. F., Monzon, F. A., Edwards, R. P. & Khan, S. A. (2008). Human papillomavirus type 16 reduces the expression of microRNA-218 in cervical carcinoma cells. Oncogene, 27(18), 2575–2582.
Palmero, E. I., de Campos, S. G. P., Campos, M., de Souza, N. C. N., Guerreiro, I. D. C., Carvalho, A. L. & Marques, M. M. C. (2011). Mechanisms and role of microRNA deregulation in cancer onset and progression. Genetics and molecular biology, 34(3), 363-370.
Peng, R. Q., Wan, H. Y., Li, H. F., Liu, M., Li, X. & Tang, H. (2012). MicroRNA-214 suppresses growth and invasiveness of cervical cancer cells by targeting UDP-N-acetyl-alpha-D-galactosamine:polypeptide N-acetylgalactosaminyltransferase 7. Journal of Biological Chemistry, 287(17), 14301-14309.
QIAGEN (15 de enero de 2013). EpiTec Methyl II Data Analysis [en red]. Recuperado de http://www.sabiosciences.com/dna_methylation_data_analysis.php
Tie, J., Pan, Y., Zhao, L., Wu, K., Liu, J., Sun, S., Guo, X., Wang, B., Gang, Y., Zhang, Y., Li, Q., Qiao, T., Zhao, Q., Nie, Y. & Fan, D. (2010). MiR-218 Inhibits Invasion and Metastasis of Gastric Cancer by Targeting the Robo1 Receptor. PLoS genetics, 6(3), 1-11.
Yamamoto, N., Kinoshita, T., Nohata, N., Itesako, T., Yoshino, H., Enokida, H., Nakagawa, M., Shozu, M. & Seki, N. (2013). Tumor suppressive microRNA-218 inhibits cancer cell migration and invasion by targeting focal adhesion pathways in cervical squamous cell carcinoma. International Journal of Oncology, 42(5), 1523-1532.
Zheng, Z. M. & Wang, X. (2011). Regulation of cellular miRNA expression by human papillomaviruses. Biochimica et Biophysica Acta, 1809(11-12), 668-677.